Activer le mode zen
Ressource au format PDF
Classification
A l'interface entre la physique et la biologie : 3 questions à Cendrine Faivre-Moskalenko.
10/12/2012
Résumé
Cet article présente les outils et les méthodes de travail de la biophysique.
Cet article appartient au dossier " 3 Questions à ..."
Table des matières
Cendrine Faivre-Moskalenko est maître de conférences au « Laboratoire de Physique », à l'Ecole Normale Supérieure de Lyon.
Elle travaille en particulier avec des montages expérimentaux issus de la physique pour étudier la dynamique d'objets biologiques.
Page personnelle :« Cendrine Faivre-Moskalenko »
1. Comment passer de la physique à la biophysique ?
Si je commence par mon parcours, j'ai fait une thèse en physique du solide à l’université de Grenoble. J’ai travaillé sur les transitions de phases de fluides confinés dans le silicium poreux, en utilisant la diffraction des rayons X, donc une technique vraiment « physique » et des questions de physique. Cependant au cours de ma thèse, j’ai eu la chance de côtoyer des gens au sein du laboratoire qui faisaient une transition vers la biophysique; thématique émergente à la fin des années 1990.
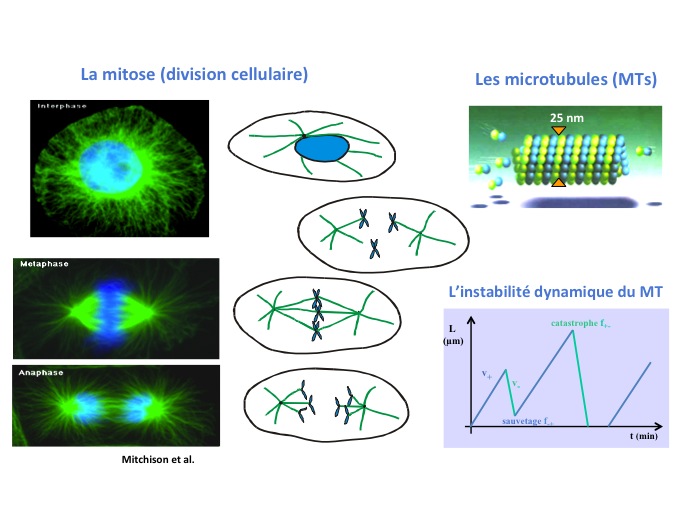
J’ai donc décidé, après ma thèse, de faire un post-doctorat à l’interface physique / biologie en ne connaissant rien de la biologie, puisque je n’avais jamais suivi de cours, mais j’étais attirée par cette thématique. Et j’ai eu la chance de partir à Amsterdam dans le laboratoire de Marileen Dogterom qui travaille sur les microtubules (figure 1), objets fascinants. J’ai été complètement et irréversiblement conquise.
Si l'on dresse maintenant un historique de cette discipline, depuis déjà très longtemps des outils physiques étaient utilisés en biologie comme la cristallographie, la RMN, la microscopie optique, qui sont des techniques développées par des physiciens mais utilisées par des biologistes. Puis, il y a eu une transition dans les années 1990 où des physiciens se sont intéressés, non plus simplement à apporter des techniques mais, eux en tant que physiciens, à travailler sur des objets biologiques. Le premier objet qui a intéressé les physiciens c’est l’ADN, comparable à un polymère, un peu spécial, mais ils l’ont étudié en tant que polymère. D'autres objets ont par la suite été étudiés par les physiciens comme : les membranes de cellules, les filaments de cytosquelette qui sont des polymères de protéines comme les microtubules (figure 1) ou l’actine qui possèdent des propriétés à la fois mécaniques et dynamiques très intéressantes et très riches.
Le microtubule est un polymère de protéines qui forme un tube creux et fait partie du cytosquelette. Dans nos cellules ce polymère est organisé sous la forme d’une étoile que l’on appelle un aster, et ce sont eux qui donnent sa rigidité à la cellule. Cet aster a un rôle important dans plusieurs phénomènes cellulaires, en particulier au moment où la cellule se divise. L'aster de microtubules est dupliqué en deux asters. Chacun migre de part et d’autres des chromosomes pour les accrocher au centre de la cellule dans le plan médian, puis vient tirer sur les chromosomes pour séparer les 2 chromatides (ou copies du génome) et former 2 cellules identiques. Le microtubule est donc un acteur à la fois de la mécanique mais aussi de la dynamique intra-cellulaire capable de générer des forces lui permettant de pousser (en polymérisant) puis tirer (en dépolymérisant) sur les chromosomes. Il est remarquable pour 2 propriétés particulières :
- La première c’est qu’il est extrêmement rigide. Sa longueur de persistance est de 5 mm. Ainsi il faudrait faire un microtubule de 5 mm pour qu’il commence à se courber sous l’effet des fluctuations thermiques. Il est 1 000 fois plus rigide que l’actine et 100 000 fois plus rigide que l’ADN.
- La deuxième propriété remarquable c’est son instabilité dynamique : il oscille entre des phases où il croît (polymérisation) et des phases où il dépolymérise. Ce mode de polymérisation est fortement hors d'équilibre. Cette croissance-décroissance permet d’aller pêcher le centre les chromosomes pour les attacher et puis ensuite en dépolymérisant d’aller tirer dessus. Il y a véritablement un lien entre la dynamique et les forces exercées par le polymère.
Il y a donc eu une transition vers l’étude des objets biologiques en tant que tels par les physiciens. Cela a aussi induit le développement de nouvelles techniques que l’on a appelées « les techniques de molécules uniques » qui ont révolutionné ce domaine de la physique. Ces techniques consistent en fait à trouver des moyens d’étudier les molécules une par une. Et parmi ces techniques de molécules uniques, il y a :
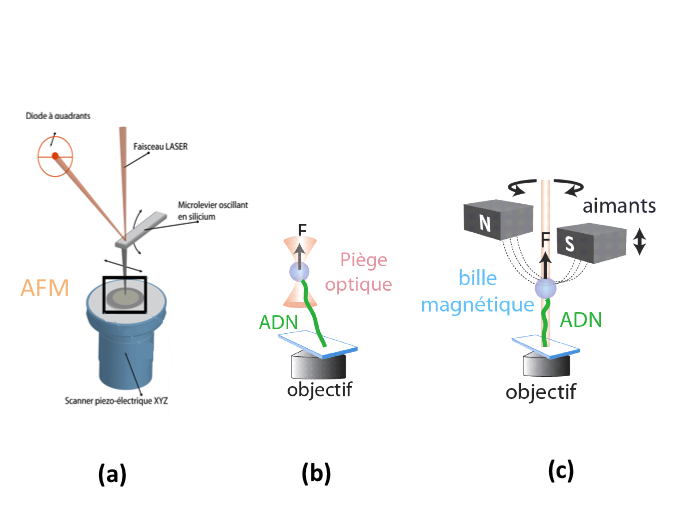
La Microscopie à Force Atomique : elle était déjà utilisée en physique du solide ou physique des surfaces pour imager un objet isolé (figure 2 (a)). En l'utilisant dans le domaine de la biophysique, on a pu visualiser : une protéine, un filament d’ADN, une membrane et exercer des forces sur ces objets. On peut, par exemple, s'amuser à déplier des protéines en utilisant le levier de l’AFM ou tirer sur l'ADN et mesurer les forces mises en jeu. Historiquement c'est la première technique qui a été appliquée pour exercer des forces sur des objets biologiques.
Les pinces optiques : c'est un système qui permet, en utilisant la lumière, de piéger des microsphères de taille de l’ordre du micron sur lesquelles on peut, par exemple, attacher un brin d’ADN pour tirer dessus (figure 2 (b)). Si on attache la bille à un moteur moléculaire tel que la kinésine, il est possible de suivre son déplacement lorsqu'il « marche » sur un microtubule.
- Les pinces magnétiques : cela consiste à utiliser un champ magnétique pour cette fois faire tourner la bille magnétique et ainsi tordre l’ADN qui est accroché dessus (figure 2 (c)). L'ADN étant une double hélice, étudier son comportement en torsion est extrêmement utile pour comprendre son interaction avec de nombreuses protéines qui agissent sur l'ADN surenroulé, par exemple les protéines topoisomérases.
Ces techniques de molécules uniques ont connu un « boom » à la fin des années 1990 et les scientifiques ont eu l’idée de les utiliser sur une diversité d’objets comme l’ADN, les protéines, les moteurs moléculaires, des complexes ADN-protéines…
En parallèle à ces techniques de micromanipulation, des techniques de fluorescence à l’échelle de la molécule unique ont été développées. On est maintenant capable de voir une seule protéine liée à un fluorophore, ce qui est révolutionnaire, et cela a permis d'apprendre beaucoup de choses sur le déplacement des moteurs moléculaires, par exemple. En effet, on est capable de les observer un par un se déplacer le long d'un rail (un microtubule par exemple) en accrochant un fluorophore dessus, alors que ce sont des objets qui mesurent seulement quelques nanomètres. Il faut réaliser que ce sont quand même des prouesses techniques incroyables !
Et puis aujourd’hui, à nouveau, on est dans une période de transition. Depuis quelques années, on change d’échelle. On a commencé à l’échelle moléculaire, puis on est passé à l'échelle de la cellule unique, et on est maintenant à l’échelle des tissus. C’est-à-dire des objets beaucoup plus complexes. On étudie la morphogénèse, où comment un organe acquiert sa forme, comment est-ce que l’on passe d’un embryon à quelque chose qui est un organisme adulte, etc. Il y a beaucoup de physique dans ces processus. Certains ont eu l'idée, par exemple, d'utiliser les concepts physiques qui décrivent le comportement des mousses de savon pour décrire la réorganisation des cellules dans un embryon. Et de nouveau, pour ces systèmes beaucoup plus complexes, de plus grande échelle, de nouvelles techniques se sont développées qui permettent de visualiser, à l'échelle nanométrique, des objets entiers comme des cellules voire des amas de cellules. Le développement de systèmes micro-fluidiques permet également de manipuler ces objets ou de les « trier » (globules rouges, ou autres cellules).
Pour revenir à une échelle plus moléculaire, on a aussi complexifié toutes les interactions entre les différents éléments que les chercheurs ont bien compris à l’échelle moléculaire, et on étudie maintenant des interactions avec des complexes multiples ADN-protéines (chromatine), l'interaction de l'ADN avec plusieurs protéines régulatrices. Et puis, comme je le fais depuis 2009, il y a aussi pas mal de choses qui se développent autour des virus, ce que l’on appelle la virologie physique, pour essayer de comprendre les étapes du cycle d’infection des virus (figure 3).
2. Comment faire le lien entre les objets inanimés de la physique vers des objets actifs ?
La diversité des objets biologiques :
C’est probablement le plus difficile justement ! Car quand on a l’habitude d’étudier des objets en physique, on calcule des barres d’erreurs et on insiste beaucoup auprès des étudiants pour qu'ils estiment les incertitudes de la mesure.
En biologie, la première chose qu’il faut apprendre, c'est qu'il n’est pas rare du tout que les barres d’erreurs fassent 100 % de la mesure. Et c’est une énorme difficulté ! Il y a une variabilité de la mesure expérimentale qui s’explique d'une part parce qu'on s’intéresse à des objets qui sont isolés, qui sont à des échelles où ils fluctuent et où, de plus, la mesure est intrinsèquement bruitée par les fluctuations thermiques, et d'autre part par une variabilité intrinsèque de beaucoup de processus biologiques liée à la complexité des partenaires et des phénomènes en jeu pour tous les processus cellulaires.
Quand on utilise des techniques de molécules uniques, par définition, on regarde un par un des objets minuscules. Et à ces échelles, tout est dominé par les fluctuations thermiques. Cela veut dire que quand on va regarder un objet et puis un autre et puis encore un autre, on peut avoir des résultats qui seront très variables, mais c’est intrinsèque à la physique de ces échelles-là. Donc tout l’enjeu est d’arriver à avoir une résolution suffisante, qu’elle soit spatiale ou temporelle, pour étudier à la fois les objets un par un pour arriver à en extraire une information quantitative, mais en même temps d'être capable de faire une mesure statistique, c’est à dire à moyenner sur un certain nombre d’objets le plus grand possible pour tirer une information qui est pertinente de la population d’objets et non pas singulière d’un objet qui pourrait se comporter différemment des autres.
Donc si l'on développe une approche expérimentale, il faut souvent pousser les instruments le plus possible pour arriver aux résolutions des toutes petites échelles. Certains chercheurs font des choses extraordinaires, ils sont capables de mesurer des déplacements nanométriques d’un moteur moléculaire sur un filament. Par contre, toute la difficulté est de savoir si c’est reproductible. Est-ce qu’on est capable de mesurer sur une population quelque chose qui est significatif de l'ensemble de la population ? Cette difficulté-là est expérimentale mais elle est aussi dans la modélisation. Il faut trouver la bonne échelle de modélisation. Si on va trop dans les détails, il va être très difficile de reproduire le phénomène mesuré avec ses incertitudes et si au contraire on fait des choses qui sont trop générales, la loi va toujours s’appliquer à une moyenne mais pas forcément décrire toute la population.
La physique statistique comme outil :
Comme les processus que l’on étudie sont à l’échelle de la molécule unique et sont par nature stochastiques, la physique statistique est un outil naturel pour étudier ces objets. Il y a un intérêt à étudier les objets isolés, à mesurer la variabilité, mais c’est aussi très important de pouvoir effectuer de nombreuses mesures pour avoir accès à la distribution statistique d’une distribution. L’information que l’on peut en tirer est beaucoup plus riche par rapport à toutes les techniques classiques utilisées pour l'étude des complexes protéiques par les biologistes ou les biochimistes qui consistent à faire des mesures d’ensemble, où l'on obtient une mesure moyenne. Dans notre cas, la moyenne d’événements doit correspondre à la mesure macroscopique, par contre on a accès à toute la distribution, et c’est dans cette distribution (dans les fluctuations) qu’il y a la richesse de l’information. La physique statistique permet ensuite d’extraire cette information via une modélisation que l’on essaie de faire la plus simple possible sur la dynamique et/ou sur la mécanique de l'objet.
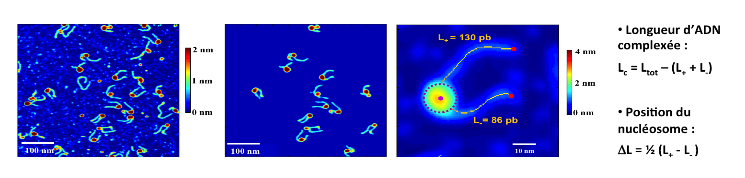
Les figures 4 et 5 illustrent les étapes nécessaires permettant de discriminer différents types de nucléosomes. Sur la figure 4, on peut voir l'image obtenue par AFM, puis un traitement numérique de l'image permet de retrouver chacun des objets pour étudier les nucléosomes un par un.
Longueur d'ADN complexée : longueur d'ADN associé aux protéines histones. Cette longueur peut fluctuer dans le temps lorsque les bras d'ADN du nucléosome se décollent des histones de manière transitoire. Le variant d'histone est une protéine qui a une séquence d'acides aminés très proche de l'histone conventionnel avec cependant quelques différences. On trouve quelques % d'histones variants dans le noyau de nos cellules.
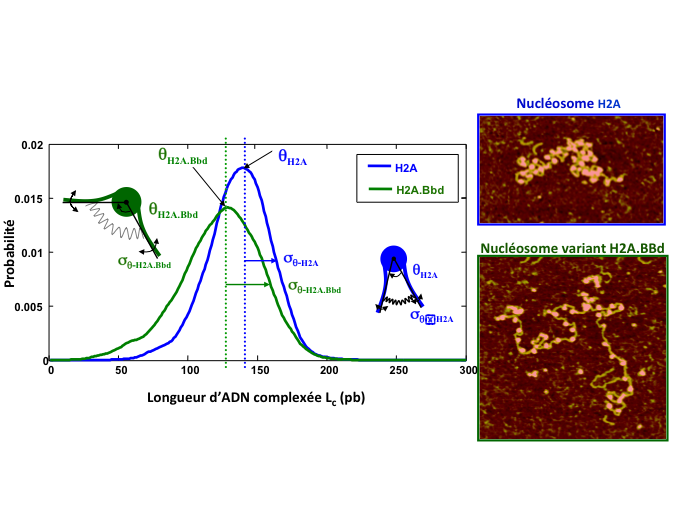
Sur la figure 5, la distribution de la longueur d'ADN complexée est tracée pour deux populations de nucléosomes. Les différentes longueurs sont représentatives de la capacité de la fibre de chromatine à se complexer.
La distribution d'ADN complexée est tracée pour plusieurs centaines de nucléosomes conventionnels H2A (en bleu) et H2A.Bbd (légèrement différent dans sa composition en protéines, en vert) et montre que le mono-nucléosome H2A est en moyenne plus complexé en ADN que le variant H2A.Bbd (valeur de θ) mais moins dynamique, l'amplitude des fluctuations σ étant réduite. Cette différence à l'échelle du mono-nucléosome a des conséquences importantes à l'échelle de la fibre de chromatine comme illustré sur les images AFM de la figure 4 : les fluctuations de longueur complexée limitent la capacité de la fibre de chromatine à se compacter, ce qui est nécessaire pour faire entrer les 2 m d'ADN contenus dans le noyau de nos cellules (environ 5 μm) ! - @ C. Moskalenko
Figure 5. Distribution d'ADN complexée
Finalement on s’aperçoit que quelque soit l’objet que l’on étudie : l’ADN avec les protéines, les nucléosomes, les microtubules, toute la dynamique de ces objets est fortement hors d’équilibre. Il y a toujours un apport d’énergie sous forme, en général, d’hydrolyse de l’ATP. Et cela fait appel à toute la physique hors d’équilibre qui est relativement récente. Il commence à y avoir nombre de théoriciens mais aussi d’expérimentateurs qui s’intéressent à des systèmes qui fluctuent. Cette nouvelle physique est particulièrement intéressante à développer aussi pour étudier ces systèmes biologiques. Et de fait, il y a un retour de la biologie vers la physique dans le sens où cette discipline offre de jolis systèmes pour les gens qui ont envie de développer de nouveaux outils de la physique hors d’équilibre. Tout l'enjeu est d'être capable de mesurer ces fluctuations, à l'équilibre ou hors d'équilibre : c'est justement ce que j'essaie de faire !
Isoler les fonctions :
On est passé de l’échelle moléculaire à l’échelle macroscopique. Cette idée d’aller décortiquer le problème et de travailler d’abord à toute petite échelle est une approche que l’on appelle « bottom-up » : on démarre du système le plus simplifié possible et on va complexifier en rajoutant un à un les éléments pour rendre le système plus réaliste.
Bien sûr, on va forcément passer par une étape qui n’intéresse pas les biologistes, parce que la toute première étape où on a un système ultra-simplifié n’est pas pertinente biologiquement. On peut aussi avoir cette critique que l'objet biologique est posé sur une surface, ou que les forces exercées ne sont pas réalistes… On est d’accord. Mais cette étape-là, c’est ce qui fait que, par la suite, en complexifiant, on est capable de découvrir la dynamique de certaines fonctions. Par exemple, sur l’ADN des expériences extraordinaires ont été réalisées et permettent de voir l’effet d’enzymes capables de couper un brin d’ADN pour dérouler le sur-enroulement et recoller ce brin. On est capable de voir le moment où l'enzyme vient agir sur l’ADN, l'endroit où elle agit, quels sont ses temps caractéristiques, combien de pas elle est capable de faire, combien de nœuds elle déroule à chaque pas, etc.
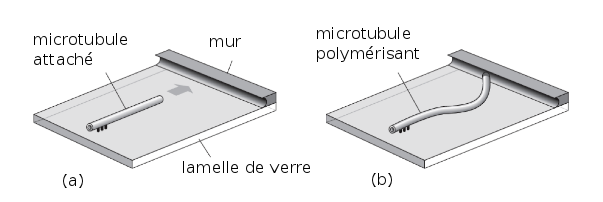
En guise d'illustration de cette approche « bottom-up », on pourrait aussi prendre l’exemple des microtubules, c’est un joli exemple. Les premières expériences réalisées par Marileen Dogterom consistaient à prendre un microtubule, à l’accrocher sur une surface d’un coté (figure 6 (a)), à mettre un mur de l'autre coté et à regarder comment il arrivait à se déformer sous l’effet de la force qu’il exerce quand il veut pousser sur le mur (figure 6 (b)). On voit une instabilité de flambage et on est donc capable de remonter à la force mécanique exercée par un filament protéique qui est en train de polymériser. On peut mesurer l’amplitude de ces forces. Ces expériences étaient évidemment irréalistes du point de vue biologique mais le fait de prouver que cette force existe, permettait de pouvoir comprendre comment les déplacements se faisaient dans la cellule.
Puis, l’étape de complexification supérieure consistait à s’intéresser à un aster de microtubules. On a été capable de regarder, cette fois, l’organisation qui ressemble plus à ce qui se passe dans la cellule. On a pu étudier comment les forces sont exercées à l’échelle globale d’un aster (figure 7). Et en complexifiant encore le système, c'est-à-dire en collant tout autour de la boite contenant l’aster des moteurs moléculaires capables de tirer, on a pu séparer la contribution des forces de poussée par la polymérisation des microtubules, et des forces de traction par les moteurs, etc.
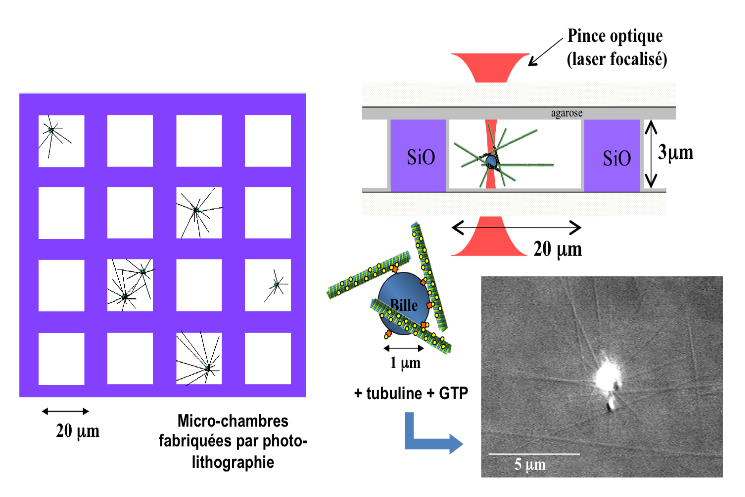
On complexifie maintenant le système en reconstruisant un aster de MTs à partir d'une bille de 1 μm qui mime le centre d'organisation des MTs (le centrosome dans la cellule) et on enferme cet aster artificiel dans une micro-chambre de 20 μm de côté qui « mime » le confinement induit par le contour d'une cellule et on observe le positionnement d'un aster au centre de la cellule uniquement induit par la force de polymérisation des MTs. On peut ensuite s'intéresser au cas plus complexe de deux asters confinés, ou encore utiliser une pince optique pour déplacer l'aster et étudier son repositionnement ou encore mesurer l'amplitude des forces générées par le positionnement. - @ C. Moskalenko
Finalement, à la fin de cette approche « bottom-up » on a un objet qui n’est évidemment pas encore celui de la cellule mais qui s'en rapproche le plus possible. Ce qu’il faut par contre réaliser c’est que l'échelle de temps pour passer de l’objet simple à l’objet très complexe, peut être supérieure à 10 ans. Complexifier le système peut s'avérer très compliqué techniquement.
On peut, au contraire, avoir une approche du type « Top-down » : en partant d’un objet très complexe, on essaie de décortiquer la contribution de chaque acteur un par un en les supprimant. Par exemple, pour savoir à quel point les forces de polymérisation des microtubules sont responsables de l’organisation de ces deux asters qui tiennent les chromosomes, on peut prendre une cellule normale, qui se divise et avec un laser détruire l'un des deux asters et regarder comment cela perturbe le bon fonctionnement cellulaire.
Les deux approches sont forcément complémentaires, on n’apprend pas la même chose. Ce qu’il faut c’est pouvoir se rejoindre quelque part au milieu des deux types d'approche pour vérifier que l'on a des résultats qui sont cohérents.
3. Quelles sont les techniques mises en jeu ?
On a parlé des techniques de micromanipulation : pinces optiques, pinces magnétiques qui permettent de manipuler des billes qui sont liées à des objets et de mesurer les forces qui s’exercent sur ces objets. Ce sont des techniques extrêmement puissantes. J'ai envie de parler d'un dernier exemple qui est l’étude du moteur moléculaire de kinésine.
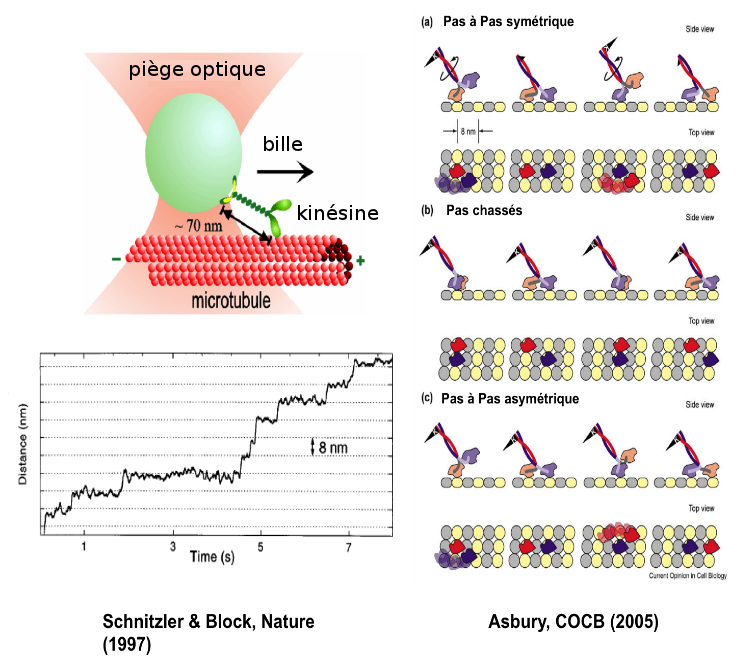
La kinésine est un moteur moléculaire qui se déplace sur les microtubules, qui transporte des vésicules (des cargos accrochés sur sa tête) et qui a deux pieds. En accrochant une grosse bille sur la tête de la kinésine, on est capable de la voir se déplacer le long d’un microtubule et de voir quel pas fait ce moteur quand la bille avance. Si on pince la bille avec une pince optique, on exerce une force sur cette bille, et on peut freiner la kinésine pour connaître la force maximum qu’on peut exercer avant qu’elle ne s’arrête. De cette façon là, les chercheurs ont pu mesurer très précisément qu’elle avançait par pas de 8 nm et qu’elle était capable de résister à une force maximum d’environ 6 pN.
Ce seul résultat est extraordinaire, mais les physiciens sont allés encore plus loin ! Ils ont voulu savoir si ces pas de 8 nm qui étaient enregistrés pour la bille, correspondaient à des pas de 16 nm ou de 8 nm pour les pieds de la kinésine, la réponse permettant de différencier les modèles de marche : pas à pas ou en pas chassés (figure 8). A l'aide de pinces optiques, ils ont montré, en faisant par exemple des mutations au niveau du corps du moteur (plus précisément de son cou) que cela la faisait « boîter » ce qui implique un mode de marche asymétrique. Ensuite, à l'aide de mesures de fluorescence à une résolution jamais égalée de l'ordre du nanomètre, ils ont été capable en accrochant un fluorophore sur un seul des 2 pieds de la protéine, de mesurer le pas du fluorophore : ils ont trouvés 16 nm. Ils en ont déduit que la kinésine marchait comme nous, en mettant un pied devant l’autre ! Ce qui est extraordinaire ! C’est tout de même un objet qui mesure entre 50 et 100 nm ! Donc ce petit moteur moléculaire se déplace comme nous et cette découverte a été permise par des techniques de molécules uniques à la fois la fluorescence et les pinces optiques, et dont la paternité revient aux physiciens !
Dans cette expérience (Schnitzer and Block, Nature - 1997), les auteurs visualisent le déplacement de la bille micrométrique par rapport au centre du piège optique en réponse à la traction exercée par la kinésine qui avance sur son rail : le MT. En observant ce déplacement en fonction du temps, on observe des marches successives de hauteur 8 nm : c'est le pas de la kinésine qui correspond exactement à la taille du maillon élémentaire du microtubule (un diamètre de tubuline). Le bruit sur la trace correspond aux fluctuations thermiques qui sont toujours présentes à cette échelle. À droite : les différents modèles de marche : pas à pas (symétrique ou asymétrique) ou pas chassé. - @ Asbury, Current Opinion in Cell Biology, 2005
J’enseigne à l’ENS l’interface physique / biologie à des étudiants en L3 et il est vrai que quand je leur parle de ces techniques de pinces optiques et que je leur raconte l’histoire de la kinésine, je vois bien que ça les fascine. L’année dernière nous avons déposé un projet pour acheter des éléments d’optiques pour monter un système de pinces optiques qui est maintenant utilisé pour les projets expérimentaux de physique. Les étudiants ont la possibilité de manipuler eux-même des billes avec la lumière. Ils ne font pas à proprement parler d'expériences de biophysique avec, parce que c’est difficile à concilier avec les contraintes d'échelles de temps des TP mais ils peuvent s'en servir pour mesurer, par exemple, des forces visqueuses exercées sur la bille.
La Microscopie à Force Atomique c’est ce que je fais tous les jours. C’est une technique d’imagerie qui donne une information topographique (donc en 3D), la contrainte est que l’on doit poser les objets sur une surface. On est capable de voir une seule molécule ADN de 2 nm de diamètre. On peut étudier la conformation de l’ADN, des nucléosomes, des virus, etc. De plus, en utilisant la pointe de l’AFM, il est possible d'appuyer sur l’objet et de mesurer la force exercée, et puisque l’on connait aussi le déplacement vertical de la pointe, d'en déduire la résistance mécanique de l’objet. Donc c’est un moyen de sonder, à la fois la morphologie mais aussi la mécanique de l’objet et c’est ce qu’on utilise pour étudier les propriétés physiques du rétrovirus VIH actuellement au laboratoire.
Récemment, il y a eu un nouvel intérêt pour la microscopie de fluorescence avec le développement de nouvelles techniques d'imagerie de super-résolution. Ces méthodes sont basées sur la détection de la position d'un fluorophore unique avec une résolution nanométrique comme l'ont développée les physiciens dans les années 2000 que l'on combine avec l'idée de fluorophores photo-activables, que l'on allumerait de manière stochastique (aléatoire) dans le temps. En effet, on a vu que l'on peut détecter très précisément l'émission de fluorescence par des molécules isolées mais cela n'est possible que si les émetteurs de photons sont suffisamment éloignés les uns des autres (au moins 200 nm, la limite de diffraction !) On utilise l'astuce qui consiste à allumer les fluorophores de façon stochastique : pour chaque image on en allume quelques uns, comme on en a peu sur chaque image on peut aller mesurer très précisément la source de l'émission. Puis en déclenchant cette excitation stochastique un certain nombre de fois, et en superposant les images, on reconstruit une image globale de l’objet, où chaque fluorophore est détecté de manière très précise, beaucoup plus précise que s’ils étaient tous allumés en même temps. Ces techniques qu'on appelle PALM, STEP, etc, permettent de faire une image de fluorescence à l'échelle d'une cellule entière avec une résolution qui est identique à celle qu'on aurait pour des objets isolés. C'est très enthousiasmant ! Pour l’instant ces méthodes ont été utilisées pour des images à très haute résolution d'une cellule ou d'un compartiment cellulaire statique mais des mesures en dynamique sont en train d'être mises au point. On va pouvoir imager directement dans la cellule des phénomènes dynamiques à une résolution spatiale jusque-là impossible.
Pour citer cet article :
A l'interface entre la physique et la biologie : 3 questions à Cendrine Faivre-Moskalenko, Cendrine Faivre-Moskalenko, décembre 2012. CultureSciences Physique - ISSN 2554-876X, https://culturesciencesphysique.ens-lyon.fr/ressource/Biophysique-Moskalenko.xml